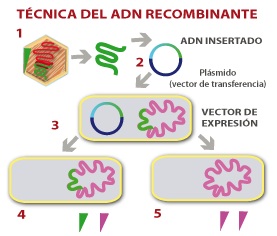
Una ejemplo de ADN recombinante,
que ocurre normalmente en la naturaleza, es la Recombinación genética. La
recombinación genética es un proceso que lleva a la obtención de un nuevo
genotipo a través del intercambio de material genético entre secuencias homólogas
de DNA de dos orígenes diferentes. Los integrones de clase 1 son elementos
genéticos que llevan un conjunto variable de genes de resistencia a
antibióticos, se propagan fácilmente de forma horizontal entre las bacterias,
lo que contribuye a la aparición de clones resistentes a múltiples fármacos. En
aislados uropatogénicos de Escherichia
coli (UPEC) que tienen integrones de clase 1, por medio de experimentos de
conjugación y transducción, las cepas donantes y resultantes se caracterizaron
por sus resistencias a los antibióticos, la presencia de genes sul1, sul2 y
sul3, matrices de casetes de integrones, replicones de plásmidos y región tra.
ADN recombinante artificial:
La construcción de proteínas de fusión como biomoléculas para el desarrollo de una vacuna viable contra la IU. Las adhesinas fimbriales (FimH, CsgA y PapG) de UPEC son factores de virulencia que contribuyen a la adhesión, invasión y formación de comunidades bacterianas intracelulares. La generación de proteínas recombinantes a partir de adhesinas fimbriales como una sola molécula se obtiene mediante tecnología de fusión. Unos pocos estudios in vivo e in vitro han demostrado que las proteínas de fusión proporcionan una respuesta inmune eficaz y protección contra la ITU producida por la UPEC.
Bibliografía:Poey ME, Laviña M. Horizontal transfer of class 1 integrons from uropathogenic Escherichia coli to E. coli K12. Microb Pathog [Internet]. abril de 2018 [citado 24 de noviembre de 2018];117:16-22. Disponible en: https://linkinghub.elsevier.com/retrieve/pii/S0882401017313578
Luna-Pineda VM, Ochoa S, Cruz-Córdova A, Cázares-Domínguez V, Vélez-González F, Hernández-Castro R, et al. Infecciones del tracto urinario, inmunidad y vacunación. Bol Med Hosp Infant Mex [Internet]. 26 de septiembre de 2018 [citado 24 de noviembre de 2018];75(2):67-78. Disponible en: http://www.ncbi.nlm.nih.gov/pubmed/29658949